导图社区 生物化学-分子生物学研究法2
- 64
- 4
- 1
- 举报
生物化学-分子生物学研究法2
关于生物化学-分子生物学研究法2的思维导图,因功能研究技术,希望这份脑图会对你有所帮助。
编辑于2023-04-24 13:14:19 山东省- 考研生物
- 生物化学与分子生物学
- 相似推荐
- 大纲
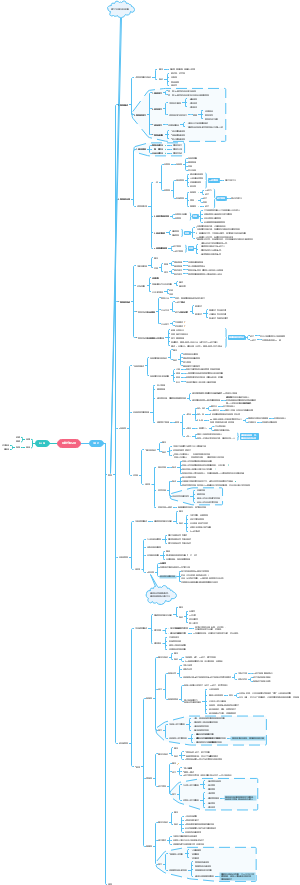
分子生物学研究法2
基因功能研究技术
基因表达研究技术
过表达
将目的基因的全长序列与高活性组成型启动子融合构建成质粒载体,通过转化,获得该基因产物大量积累
转录组测序和RNA-Seq
转录组:某一特定生理条件或环境下,一个细胞、组织或生物体中所有RNA的总和,包括mRNA、tRNA、rRNA及非编码RNA;狭义上指细胞中转录出来的所有mRNA的总和。基因组-转录组-蛋白质组是中心法则在组学框架下的主要表现形式 - **基于传统的Sanger测序法对转录组进行研究的方法**:*表达序列标签技术(EST)*;*基因表达系列分析技术(SAGE)*。EST测序数据----数据最多,涉及物种最广的转录组数据/ 测序读长较短,通量小,成本高 - 高通量测序技术 可以一次性测序几十万~上百万条序列 - RNA-Seq:利用高通量测序技术对转录组进行测序分析,对测序得到的大量原始读长进行过滤、组装和生物信息学分析的过程。 对于由参考基因组序列的物种,需要根据参考序列进行组装;没有参考序列的,需要从头进行组装,利用大量读长之间重叠覆盖和成对读长的相对位置关系,组装得到尽可能完整的转录本,并以单位长度转录本上覆盖的读长数目作为衡量基因表达水平的标准。 基于二代测序的RNA-Seq读长较短,得到的基因转录产物通过一定的算法进行组装得到的,可能会导致较多的嵌合体组装错误,对后续的转录组分析造成影响;基于PacBio单分子实时测序的三代测序法,超长读长可以精确进行全长转录组测序,建库过程中无需打断转录本
RNA的选择性剪接研究
RNA-Seq可用于研究全基因组水平的RNA选择性剪接。RNA的选择性剪接:用不同的剪接方式从一个mRNA前体产生不同mRNA剪接体的过程 分类:内含子保留、3'/5'选择性剪接;外显子遗漏型剪接;相互排斥性剪接 常用**RT-PCR**的方法在*单个基因水平*上*验证由RNA-Seq预测得到的选择性剪接的真实性*:以*cDNA两端特异性引物*或来自*不同外显子的引物序列* 扩增不同组织来源的RNA样品,观察PCR产物是否存在差异。如果存在差异,测序后检测这种差异是否来自于选择性剪接。
基因定点突变技术
- 通过**改变基因特定位点核苷酸序列从而改变编码的氨基酸序列**,用于研究某个氨基酸残基对蛋白质的结构、催化活性及结合配体能力的影响,也可用于改造DNA调控原件特征序列、修饰表达载体、引入新的酶切位点等 - PCR介导的定点突变方法(主要) - 亚克隆 - T/A克隆 - 加限制性酶切位点 - 平端连接 - 产生DNA探针 - PCR的诊断应用 - - - PCR介导的方法的优点: - 突变体回收率高,有时不需要进行突变体筛选 - 能用双链DNA作为模板,可以在任意位点引入突变 - 可以在同一试管中完成所有反应 - 快捷简便,无需在噬菌体M13载体上进行分子克隆
基因表达检测技术
研究单个或多个基因在生物体内某些特定发育阶段或在不同环境条件下的表达模式
基因芯片技术
原位杂交技术
用标记的核酸探针,经放射自显影或非放射检测体系,在组织、细胞、间期核和染色体上对核酸进行定位和相对定量研究的一种方法,通常分为**RNA原位杂交**和**染色体原位杂交**两类。 原理:**RNA原位杂交**用*放射性*或*非放射性*标记的**特异性探针**与被**固定的组织切片**反应,若细胞中存在与探针互补的*mRNA分子*,两者经杂交产生**双链RNA**,就可通过**检测放射性标记**或**酶促免疫显色**,对该基因的表达产物在细胞水平上做出**定性定量**分析。 **荧光原位杂交(FISH)** 对寡核苷酸探针做特殊的修饰和标记,然后用原位杂交法与靶染色体或DNA上特定的序列结合,在通过荧光分子偶联的单克隆抗体确定该DNA序列在染色体上的位置 优点:不需要放射性同位素,实验周期短,检验灵敏度高,可在同一张切片上观察几种不同的DNA探针的定位,得到相应位置和排列顺序的综合信息。
转录组测序技术
基因敲除技术
基本原理
通过外源DNA与染色体DNA之间的同源重组,进行精确的定点修饰和基因改造,具有专一性强、染色体DNA可与目的片段共同稳定遗传等特点。
完全基因敲除
通过同源重组法完全消除细胞或动植物体中靶基因活性 - 实验中一般采用取代型或插入型载体在ES细胞中根据正-负双向选择(PNS)原理进行完全基因敲除实验 - **正向选择基因\***neo\*通常被*插入*载体靶DNA功能最关键的**外显子**中,或通过同源重组法*置换靶基因的功能区* - 负向选择基因*HSV-tk*被置于目的片段外侧,含有该基因的重组细胞不能在选择培养基上生长。如果细胞中发生了随机重组,负向选择基因可能被整合到基因组中,导致细胞死亡
条件型基因敲除
对于许多有重要生理功能的基因而言,完全基因敲除往往导致胚胎死亡,有关该基因的功能研究无法开展 构建条件型基因敲除打靶载体时,常常将正向选择基因**neo**置于靶基因*内含子*中,并在*靶基因重要功能域两侧内含子中*插入方向相同的**LoxP**位点。当实验需要消除靶基因活性时,与带有**Cre重组酶**基因的ES细胞杂交,Cre重组酶就能将两个LoxP位点中间的DNA片段切除,导致靶基因失活。 通过定位重组系统实现特定时间和空间的基因敲除
PAS法筛选已发生同源重组的细胞
基因捕获法
高等动物基因敲除技术
- 电穿孔 - 显微注射 将重组DNA导入胚胎干细胞纯系中,使外源DNA与胚胎干细胞基因组中相应部位发生同源重组,将重组载体中DNA序列整合到内源基因组中并得以表达
植物基因敲除技术
- T-DNA插入失活技术 利用农杆菌T-DNA介导转化,将带有报告基因的DNA序列整合到基因组DNA上,如果这段DNA到目的基因内部或附近,就会影响该基因的表达,从而使该基因失活
基因组编辑技术
利用序列特异核酸酶在基因组特异位点产生DNA双链断裂,从而激活生物体自身的同源重组或非同源重组末端连接修复机制,以达到特异性改造基因的目的 基因编辑工具: - 锌指核酸酶 - TALEN - CRISPR-Cas9 - 该系统原为细菌及古菌适应性免疫系统的一部分,其功能为抵御病毒及外源DNA的入侵。CRISPR-Cas9系统由CRISPR序列和Cas基因家族组成。CRISPR序列是高度保守的正向重复短回文序列,Cas基因簇位于CRISPR序列的5'端,编码的蛋白质可切割外源DNA。 - CRISPR/Cas系统分为I型、II型和III型。II型应用最为广泛。外源基因入侵时,CRISPR序列转录并被加工为约40nt的成熟crRNA(CRISPR RNAs)。 - crRNA与tracrRNA通过碱基互补配对形成双链RNA,激活并引导Cas9切割外源DNA中原型间隔序列(protospacer)。 - Cas9蛋白具有2个核酸酶结构域:RuvC-like结构域(切割非互补链)和HNH结构域(切割原型间隔序列中与crRNA互补配对的DNA链);Cas9对靶序列的编辑依赖于原型间隔序列下游的短序列PAM,PAM通常为5'-NGG-3';极少数为5'-NAG-3'。Cas9的切割位点位于PAM上游的第三个碱基 - 将crRNA与tracrRNA双分子结构融合成具有发夹结构的sgRNA(single guide RNA),sgRNA分子5'端20nt的引导序列完全与DNA靶序列互补,从而引导Cas9对靶DNA进行编辑。只要改变sgRNA上20nt的引导序列,基因组上任意5'-(N)20-NGG-3'序列都可被CRISPR-Cas9编辑
蛋白质及RNA互作技术(功能调控及检测)
酵母单杂交系统
可识别稳定结合于DNA上的蛋白质,可在酵母细胞内研究真核生物中DNA-蛋白质之间的相互作用,并通过筛选DNA文库直接获得靶序列相互作用的蛋白的编码基因;分析鉴定细胞中转录调控因子与顺式作用元件相互作用的有效方法 **基本原理:**首先将已知的特定顺式作用元件构建到基本启动子上游,把报告基因构建到基本启动子下游。然后,将编码待测转录因子cDNA与已知酵母转录激活域(AD)融合表达载体导入酵母细胞,该基因产物如果能与顺式作用元件结合,就能激活基本启动子,使报告基因表达 **作用:** - 某个DNA分子与某个蛋白质分子是否相互作用 - 用于分离*编码结合于特定顺式作用元件或其他DNA位点的*功能蛋白编码基因 - 验证反式转录调控因子的的DNA结合结构域,准确定位参与特定蛋白质结合的核苷酸序列 - 克隆细胞中含量极低且用生化手段难以纯化的那部分转录调控因子
酵母双杂交系统
DNA结合域和转录激活结构域(AD)是转录激活因子发挥功能所必需。 - 单独的BD能与特定基因的启动区结合,但不能激活基因转录;由不同转录调控因子的BD和AD形成的杂合蛋白能行驶激活转录的功能 - 实验中,首先运用基因重组技术把**编码已知蛋白的DNA序列**连接到带有**酵母细胞转录调控因子BD结构域基因片段的表达载体**上。导入酵母细胞使之表达带有DNA结合结构域的杂合蛋白,与报告基因上游的启动调控区相结合,准备作为“**诱饵**”捕获与已知蛋白相互作用的基因产物。 - 将已知的**AD的基因片段**与**待筛选的cDNA文库中不同插入片段**相连接,获得**猎物载体** - **转化**含有“诱饵”的酵母细胞,*只要酵母细胞中表达的“诱饵蛋白”与“猎物”载体中表达的某个蛋白发生相互作用*,不同转录调控因子的***AD与BD结构域就会牵引靠拢,激活报告基因表达***。
蛋白质相互作用技术
等离子表面共振技术
**原理** - 将诱饵蛋白结合于葡聚糖表面,将葡聚糖层固定于纳米级厚度的金属膜表面。 - 当由蛋白质混合物经过时,如果有蛋白质与诱饵蛋白发生相互作用,那么二者的结合使金属膜表面的折射率上升,从而导致共振角度的改变 - 共振角度的改变与该处蛋白质浓度呈线性关系,由此可检验蛋白质之间的相互作用 优缺点 - 不需要标记物和燃料 - 安全灵敏快速,可定量分析 - 需要专门的等离子表面共振检测仪器
免疫共沉淀技术
通过抗体特异性识别抗体蛋白 **原理** - 通过亲和反应,将**靶蛋白的抗体**连接到**固体基质**上 - 将可能与靶蛋白相互作用的待**筛选蛋白**加入*反应体系* - *低离心力*沉淀或微膜过滤法在固体基质和抗体的共同作用下将蛋白质复合物沉淀到试管底部或微膜上 - 如果*靶蛋白与待筛选蛋白发生了相互作用*,则**该抗体蛋白可通过靶蛋白与抗体和固体基质的相互作用而被分离**
GST及GAD融合蛋白沉降技术
利用**GST对谷胱甘肽偶联的琼脂糖球珠的亲和性**,从混合蛋白质样品中纯化得到相互作用的蛋白 GST沉降试验通常有2种应用:确定探针蛋白与未知蛋白间的相互作用;确证探针蛋白与某个已知蛋白的之间的相互作用
荧光共振能量转移
染色质免疫共沉淀技术(ChIP)
研究活体细胞内染色体DNA与蛋白质相互作用的技术 **主要流程**: - 在活细胞状态下固定蛋白质-DNA复合物,并通过超声或酶处理将其随机切断为一定长度的染色质小片段 - 通过抗原抗体的特异性反应,沉淀该复合物,从而富集与目的蛋白相结合的DNA片段 - 对目的片段进行纯化和测序分析,就能获知与目的蛋白发生相互作用的DNA序列信息(包括DNA序列特征、在基因组上位置、结合亲和程度及对基因表达的影响) **作用**: - 检测体内**转录调控因子与DNA的动态作用** - 研究组蛋白的各种共价修饰与基因表达的关系 - 定性或定量检测体内转录因子与DNA的动态作用
RNAi技术及其应用
- 利用**双链小RNA** 高效、特异性**降解细胞内同源mRNA**从而**阻断基因表达**,使细胞出现**靶基因缺失**的表型 - 双链RNA是RNAi的引发物,引发与之互补的单链RNA的降解。经过Dicer酶的加工细胞中较长的双链RNA被降解为21~25个核苷酸的小分子干扰核糖核酸,并有效定位目标mRNA - RNAi作用机制 - 由siRNA中反义链参与指导合成RNA诱导的沉默复合体(RISC),再由RISC介导切割目的mRNA分子中与siRNA反义链互补的区域,从而实现干扰靶基因表达的功能。 - -
在酵母细胞中鉴定靶基因功能
酵母基因转化与性状互补
外源基因在酵母中的功能鉴定
其他分子生物学技术
凝胶滞缓技术 噬菌体展示技术 蛋白质磷酸化分析技术 蛋白质免疫印记实验 细胞定位及染色技术 全基因组关联分析及应用